注释数据集的产生和可用性对于自动化表型分析方法的训练和评估是必不可少的。由于基于3D视觉的表型分析技术的进步以及难以完全注释复杂的3D植物结构,对具有器官级标记的真实植物的完整3D模型的需求更加明显。
本文介绍了用X射线断层成像技术获得的11个带注释的真实蔷薇属植物3D模型的ROSE-X数据集。手动执行注释,以便以器官标签的形式为与植物茎相对应的体素提供地面真实数据。此数据集既可作为执行器官级分割的监督学习方法的训练数据,也可作为评估其性能的基准。数据集中的rosebush模型具有高质量和复杂的结构,器官之间经常互相重叠交叉对现有的植物器官分割方法提出了挑战。作者报告了使用四种基线方法获得的叶/茎分割结果。通过使用随机森林分类器训练局部特征的体积方法可获得最佳性能,叶子和茎类的联合相交(IoU)值分别为97.93%和86.23%。
数据集中的rosebush模型示例。对原始的X射线体积进行阈值处理和掩码处理,得到如图a所示的实体部分。体积中的每个体素被注释为叶、茎、花、盆或标签,以获得如b所示的地面真实分割。c中,只显示了植物地上部分,不包括花盆和标签。d中显示了只与茎类对应的体素。
通过基线方法预测的玫瑰花灌木样本的叶和茎标签。
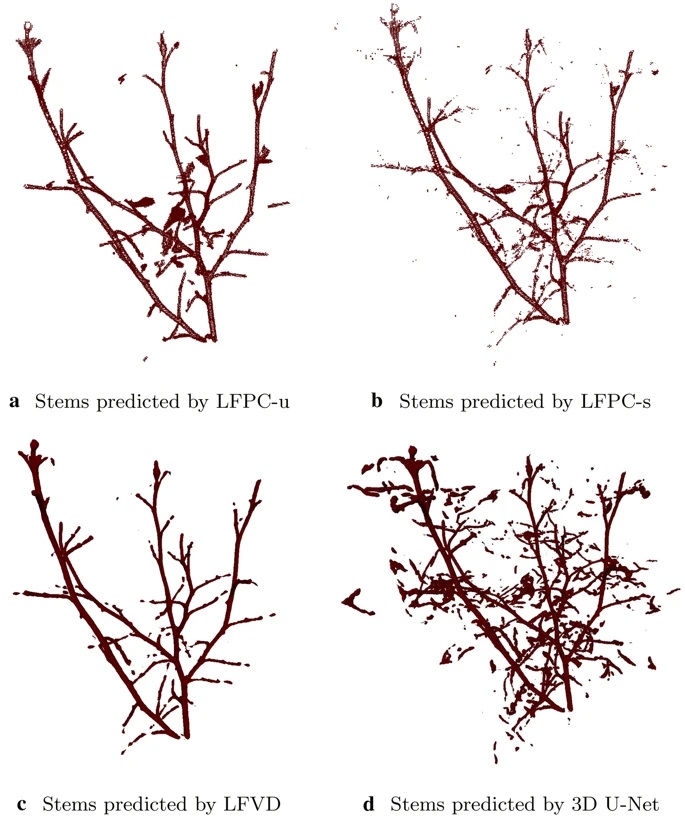
通过基线方法预测的玫瑰花灌木样本的茎标签。

红色椭圆突出显示的基线方法的错误预测示例。
作者提供了11种蔷薇属植物的带注释3D数据集,用于器官分割方法的训练和评估。还报告了基线方法的叶/茎分割结果,这些结果有待改进。该数据集与基线结果一起,有可能成为未来植物自动表型研究的重要资源。
来源:Plant Methods.ROSE-X: an annotated data set for evaluation of 3D plant organ segmentation methods.Helin Dutagaci, Pejman Rasti, Gilles Galopin & David Rousseau.https://plantmethods.biomedcentral.com/articles/10.1186/s13007-020-00573-w